综合介绍
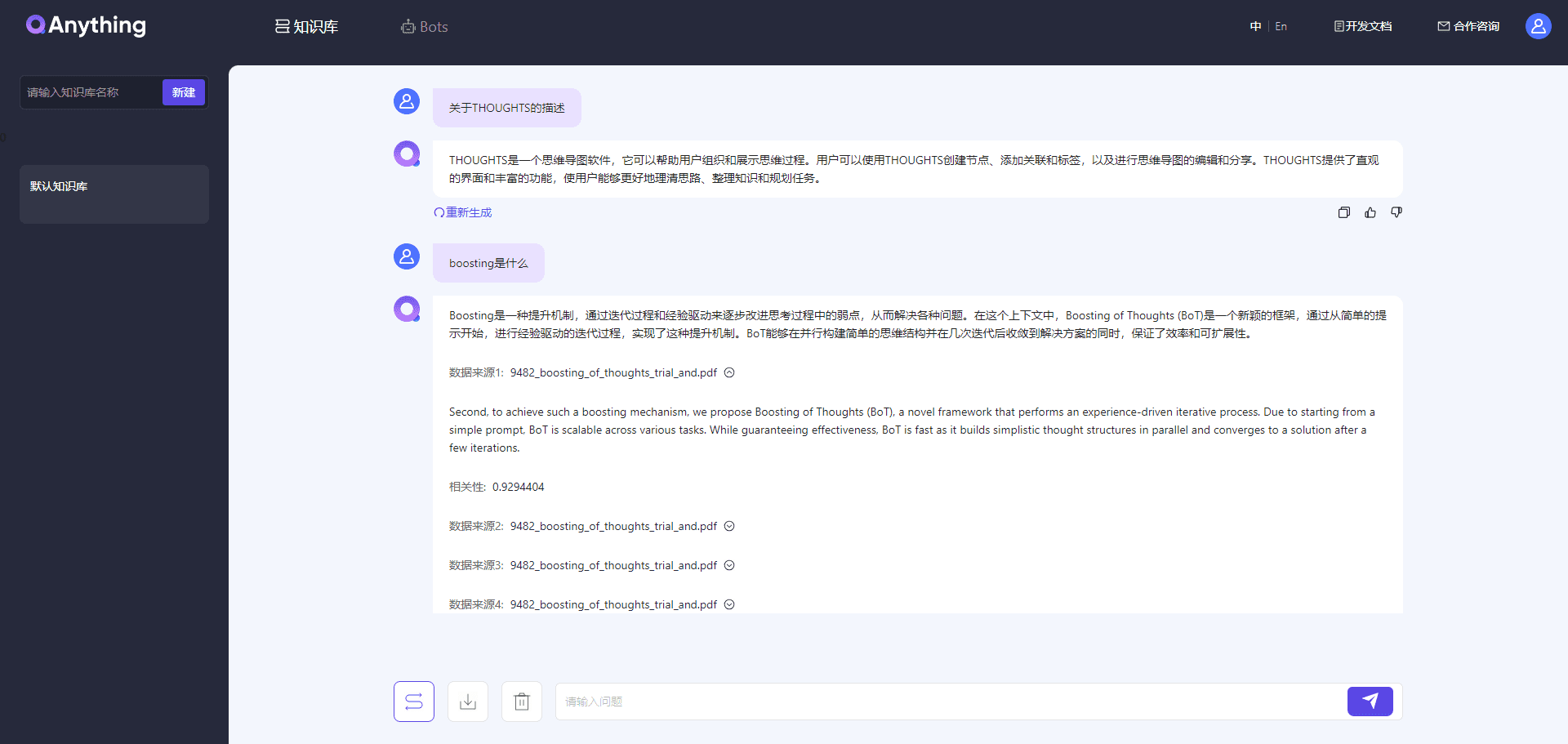
SciToolAgent 是由杭州浙江大学创新中心(HICAI-ZJU)开发的一个开源工具平台。它通过知识图谱(SciToolKG)和大语言模型技术,整合超过500种科学工具,帮助科研人员处理生物、化学、材料科学等领域的问题。这个工具的主要用途是自动化完成科研任务,比如数据处理、分子建模或文献分析,最终生成具体成果,如分析图表、计算结果或总结报告。平台免费托管在 GitHub 上,用户可以本地部署或通过在线服务体验,特别适合需要快速产出成果的科研团队。

功能列表
- 工具整合:连接500多种工具,处理数据、建模或计算任务。
- 知识图谱推荐:根据任务需求,推荐合适的工具组合。
- 任务自动化:自动规划并执行科研流程,节省时间。
- 成果生成:输出图表、报告或计算结果等具体成果。
- 安全监控:确保工具运行安全,避免错误结果。
- 自定义扩展:支持添加新工具,产出个性化成果。
使用帮助
安装流程
SciToolAgent 需要本地安装才能完整使用。步骤如下:
1. 下载源码
- 在终端输入:
git clone https://github.com/HICAI-ZJU/SciToolAgent.git
cd SciToolAgent
2. 创建虚拟环境
- 创建并激活环境:
conda create -n SciToolAgent python=3.10
conda activate SciToolAgent
3. 安装依赖
- 安装完整依赖:
pip install -r requirements.txt
- 或安装精简版:
pip install -r requirement_agent.txt
4. 配置环境
- 复制并编辑
.env文件:
OPENAI_API_BASE = your_api_base
OPENAI_API_KEY = your_api_key
5. 启动服务
- 进入
tools目录,运行:
cd tools
bash run.sh
如何使用
SciToolAgent 可以通过本地运行或在线体验两种方式输出成果。
本地运行
- 启动服务:运行
bash run.sh后,服务在本地待命。 - 执行任务:进入
test目录,运行测试脚本:
cd ../test
PYTHONPATH=. python test_run_SciToolAgent.py
- 查看成果:任务完成后,结果保存在
tools/TempFiles,如图表或报告。 - 运行案例:根目录
Cases.ipynb提供四个示例,可直接运行查看输出。
在线体验
- 访问在线服务:
http://scitoolagent.scimind.ai:8080/ - 输入任务(如“分析分子数据”),提交后几秒内返回简单成果。
- 在线版适合快速测试,复杂成果需本地运行。
特色功能操作流程
1. 数据分析并输出图表
- 用途:处理数据并生成可视化成果。
- 步骤:
- 将数据文件(如
.csv)放入tools/DataFiles。 - 运行命令:
python scripts/run_agent.py --input "tools/DataFiles/sample.csv" --task "数据分析并可视化"
- 检查
tools/TempFiles,获取生成的图表和分析结果。
2. 分子建模生成计算结果
- 用途:分析分子结构并输出计算数据。
- 步骤:
- 准备分子文件(如
.cif),放入tools/DataFiles。 - 调用工具:
from tools.ToolsFuns import molecular_analysis
result = molecular_analysis("tools/DataFiles/input_file.cif")
print(result)
- 输出结果为分子参数或模型数据,保存在指定路径。
3. 自动化生成科学报告
- 用途:整合多工具输出完整报告。
- 步骤:
- 输入任务描述和数据文件:
python scripts/run_agent.py --input "tools/DataFiles/sample.pdf" --task "分析文献并生成报告" - 系统自动调用工具,处理文献并生成总结。
- 报告保存在
tools/TempFiles,格式为文本或PDF。
注意事项
- 本地运行需16GB内存和稳定网络,确保复杂任务顺利完成。
- 在线版适合简单成果,数据量大时建议本地部署。
- 定期清理
tools/TempFiles,避免占用过多空间。
© 版权声明
文章版权归 AI分享圈 所有,未经允许请勿转载。
相关文章

暂无评论...