Protenix-v1是什么
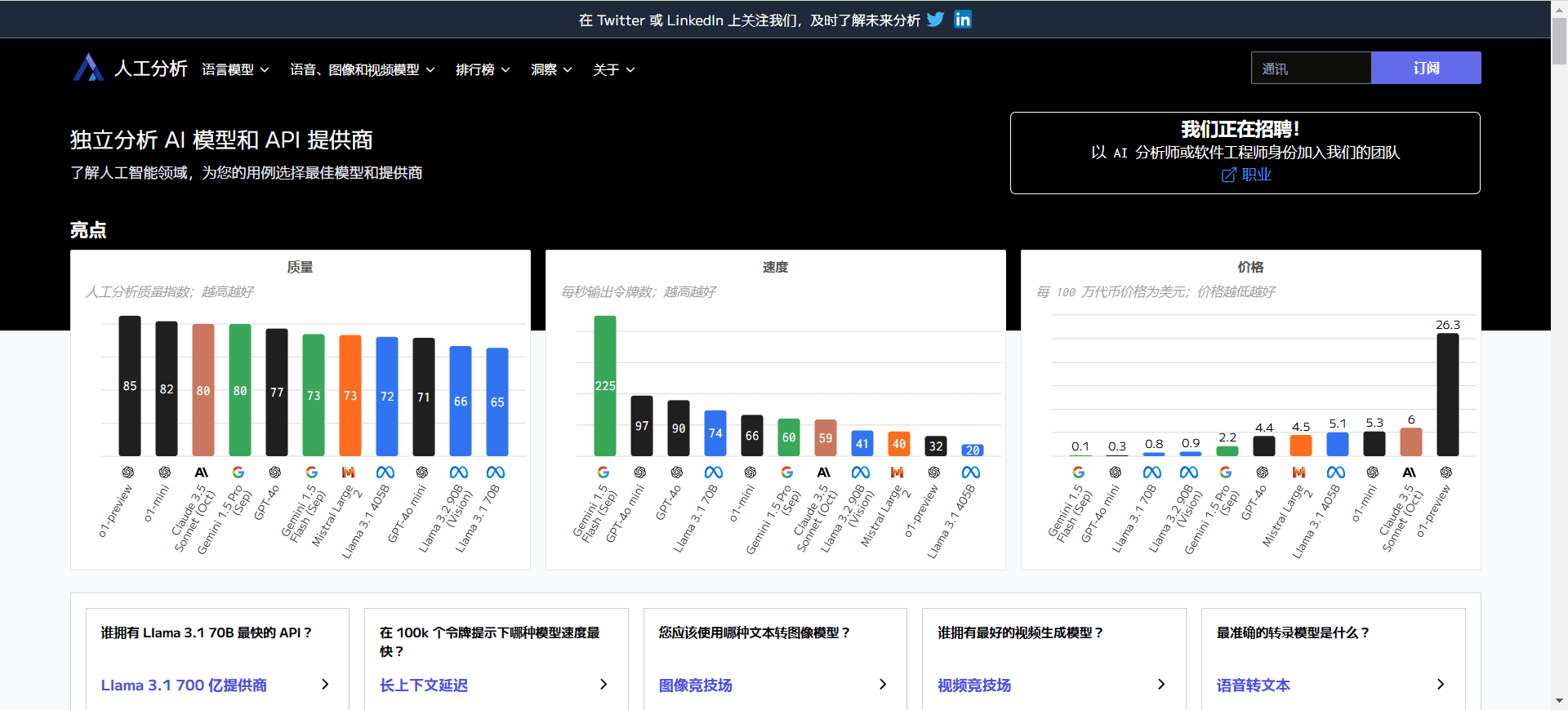
Protenix-v1是字节跳动ByteDance Seed团队推出的首个开源蛋白质结构预测模型,性能在严格对齐训练数据和模型规模后超越AlphaFold 3。模型具备显著的推理时扩展特性:通过增加采样次数(如随机种子数),预测精度可近似线性提升,例如在抗体-抗原任务中,采样种子从1个增至80个时,DockQ成功率从36%提升至48%。Protenix-v1支持蛋白质、RNA、DNA及小分子的复合物预测,并推出双版本策略——严格对齐AF3数据的版本用于学术研究,2025版则整合最新数据提升工业应用价值。

Protenix-v1的功能特色
- 全原子生物分子结构预测:支持蛋白质、核酸(DNA/RNA)以及小分子配体等复杂生物系统的全原子3D结构预测。
- 蛋白质-蛋白质复合物预测:精准预测蛋白质相互作用形成的复合物三维结构。
- 抗体-抗原复合物预测:专门优化抗体与抗原结合界面的结构预测能力。
- 核酸结构预测:支持DNA和RNA分子的三维结构建模。
- 小分子配体结合预测:可预测小分子药物与靶点蛋白的结合构象。
- 推理时Scaling能力:支持通过增加采样候选数量(从几个到数百个)实现近似对数线性的准确率提升。
- 双版本策略:提供标准版(数据截止2021-09-30,用于学术对比)和实战版(数据截止2025-06-30,用于药物发现)。
- 配套设计工具PXDesign:基于Protenix的binder设计套件,支持从头设计结合蛋白。
- 轻量级变体Protenix-Mini:大幅降低推理成本,同时保持较高精度。
- Web Server在线服务:提供基于浏览器的交互式预测平台,无需本地部署。
Protenix-v1的核心优势
- 全原子3D结构预测:能精准处理蛋白质、核酸(DNA/RNA)以及小分子配体等复杂生物系统。
- 严格公平对比:训练数据截止2021年9月30日(与AF3一致),模型参数量368M,确保与AlphaFold 3的公平比较。
- 性能超越AF3:在FoldBench修正基准集上,蛋白质-蛋白质及抗体-抗原复合物预测均优于AlphaFold 3,大幅领先Chai-1、Boltz-1等开源模型。
- 推理时Scaling能力:支持通过增加采样候选数量(从几个到数百个)实现近似对数线性的准确率提升,抗体-抗原预测DockQ成功率可从36%提升至47.68%。
Protenix-v1官网是什么
- GitHub仓库:https://github.com/bytedance/Protenix
- 技术论文:https://github.com/bytedance/Protenix/blob/main/docs/PTX_V1_Technical_Report_202602042356.pdf
Protenix-v1的适用人群
- 结构生物学家:需要预测蛋白质、核酸等生物大分子三维结构的研究人员。
- 计算生物学家:从事生物分子建模、分子动力学模拟等领域的科研人员。
- 药物研发科学家:进行靶点发现、药物设计、分子对接的制药行业从业者。
- 抗体工程研究人员:专注于抗体-抗原相互作用预测和抗体优化的研发团队。
- AI for Science研究者:探索深度学习在生命科学领域应用的学术和工业界研究人员。
- 生物信息学工程师:需要集成结构预测工具到分析流程的技术人员。
© 版权声明
文章版权归 AI分享圈 所有,未经允许请勿转载。
相关文章

暂无评论...