SciToolAgent: uma inteligência que integra mais de 500 ferramentas de pesquisa para automatizar tarefas científicas e de pesquisa
Introdução geral
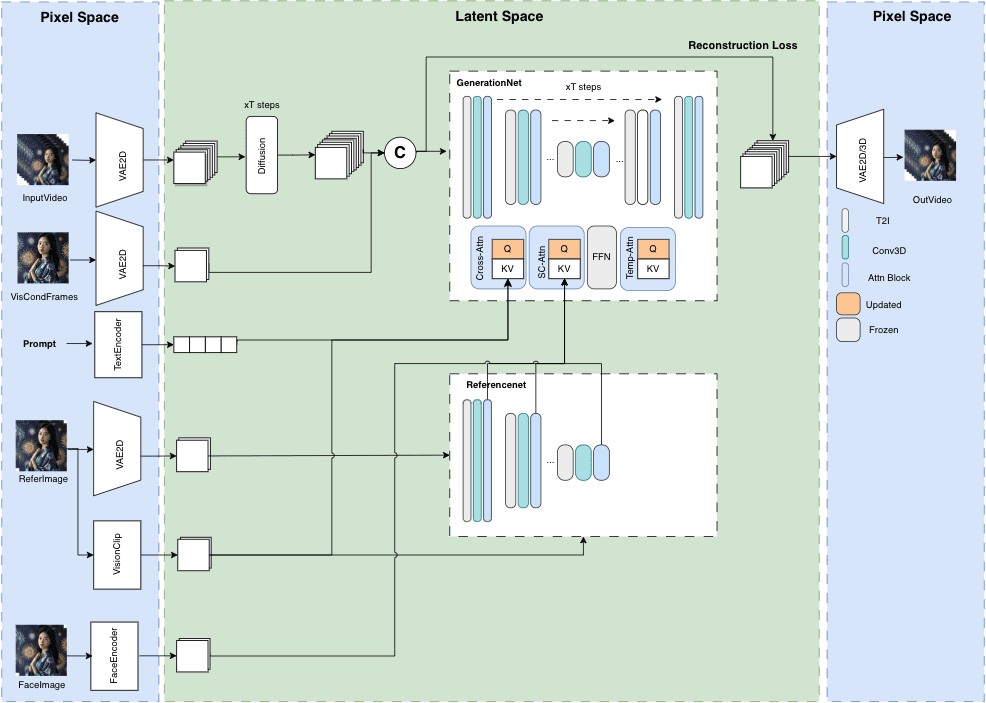
O SciToolAgent é uma plataforma de ferramentas de código aberto desenvolvida pelo Centro de Inovação da Universidade de Zhejiang em Hangzhou (HICAI-ZJU). Ela integra mais de 500 ferramentas científicas por meio de gráficos de conhecimento (SciToolKG) e tecnologias de modelagem de big language para ajudar os pesquisadores a lidar com problemas nas áreas de biologia, química, ciência dos materiais e assim por diante. O principal uso dessa ferramenta é automatizar tarefas científicas, como processamento de dados, modelagem molecular ou análise de literatura, e, por fim, gerar resultados específicos, como gráficos analíticos, resultados computacionais ou relatórios resumidos. A plataforma é hospedada gratuitamente no GitHub e pode ser implantada localmente ou experimentada por meio de um serviço on-line, o que a torna particularmente adequada para equipes de pesquisa que precisam produzir resultados rapidamente.

Lista de funções
- Integração de ferramentasConecte-se a mais de 500 ferramentas para lidar com dados, modelagem ou tarefas de cálculo.
- Recomendações do gráfico de conhecimentoRecomendação: Recomendar a combinação certa de ferramentas com base nas necessidades da tarefa.
- Automação de tarefasEconomize tempo planejando e executando automaticamente os processos de pesquisa.
- Geração de resultadosSaída de resultados específicos, como gráficos, relatórios ou cálculos.
- monitoramento de segurançaGarantia de que a ferramenta seja executada com segurança e evite resultados errôneos.
- Extensões personalizadasSuporte à adição de novas ferramentas para produzir resultados personalizados.
Usando a Ajuda
Processo de instalação
O SciToolAgent requer uma instalação local para ser totalmente funcional. As etapas são as seguintes:
1. download do código-fonte
- Digite-o no terminal:
git clone https://github.com/HICAI-ZJU/SciToolAgent.git
cd SciToolAgent
2. criação de ambientes virtuais
- Crie e ative o ambiente:
conda create -n SciToolAgent python=3.10
conda activate SciToolAgent
3. instalação de dependências
- Instale a dependência completa:
pip install -r requirements.txt
- Ou instale a versão lite:
pip install -r requirement_agent.txt
4. ambiente de configuração
- Copiar e editar
.envDocumentação:
OPENAI_API_BASE = your_api_base
OPENAI_API_KEY = your_api_key
5. ativação de serviços
- entrar em
toolsCatálogo, executado:
cd tools
bash run.sh
Como usar
O SciToolAgent pode gerar resultados executando-os localmente ou experimentando-os on-line.
operação local
- Início dos serviços: Executar
bash run.shDepois disso, o serviço fica em espera localmente. - operar: Acesso
testexecute o script de teste:
cd ../test
PYTHONPATH=. python test_run_SciToolAgent.py
- Exibir resultadosApós a conclusão da tarefa, os resultados são salvos na pasta
tools/TempFilescomo gráficos ou relatórios. - caixa de corrida: Diretório raiz
Cases.ipynbQuatro exemplos são fornecidos e podem ser executados diretamente para visualizar o resultado.
Experiência on-line
- Acesso a serviços on-line:
http://scitoolagent.scimind.ai:8080/ - Insira uma tarefa (por exemplo, "Analisar dados moleculares") e retorne resultados simples em segundos após o envio.
- A versão on-line é adequada para testes rápidos, mas os resultados complexos precisam ser executados localmente.
Funções em destaque Procedimento de operação
1. análise de dados e saída de gráficos
- usoProcessamento de dados e geração de visualizações.
- mover::
- Para converter um arquivo de dados (por exemplo
.csv(computação) colocar (em)tools/DataFiles. - Executar comando:
python scripts/run_agent.py --input "tools/DataFiles/sample.csv" --task "数据分析并可视化"
- sonda
tools/TempFilesA seguir, alguns exemplos de gráficos e análises que podem ser usados para gerar os resultados.
2. modelagem molecular para gerar cálculos
- usoAnálise de estruturas moleculares: analisa estruturas moleculares e gera dados calculados.
- mover::
- Preparar documentos moleculares (por exemplo
.cif), insira otools/DataFiles. - Ferramentas de chamada:
from tools.ToolsFuns import molecular_analysis
result = molecular_analysis("tools/DataFiles/input_file.cif")
print(result)
- Os resultados de saída são parâmetros moleculares ou dados do modelo, salvos no caminho especificado.
3. geração automatizada de relatórios científicos
- usoIntegração de várias ferramentas para gerar relatórios completos.
- mover::
- Insira a descrição da tarefa e o arquivo de dados:
python scripts/run_agent.py --input "tools/DataFiles/sample.pdf" --task "分析文献并生成报告" - O sistema invoca automaticamente a ferramenta, processa a literatura e gera um resumo.
- O relatório é mantido no
tools/TempFilesO formato é texto ou PDF.
advertência
- A operação local requer 16 GB de RAM e uma rede estável para garantir a conclusão bem-sucedida de tarefas complexas.
- A versão on-line é adequada para resultados simples, e a implantação local é recomendada para grandes quantidades de dados.
- Limpeza regular
tools/TempFilespara não ocupar muito espaço.
© declaração de direitos autorais
Direitos autorais do artigo Círculo de compartilhamento de IA A todos, favor não reproduzir sem permissão.
Artigos relacionados

Nenhum comentário...